- 国内站
- 国际站
无数据


近日,美国加州理工学院钱璐璐(Lulu Qian)教授课题组在2025年9月3日、10月1日分别以题为“Supervised learning in DNA neural networks”和“Heat-rechargeable computation in DNA logic circuits and neural networks” 发表2篇Nature研究论文。2篇论文作者都只有2人,钱璐璐教授是唯一通讯作者。2篇论文第一作者分别是Kevin M. Cherry博士(2023-2024 博士后),Tianqi Song 博士(现任教职于UNC Greensboro助理教授)。两项研究解决了分子计算领域长期存在的能源供给和学习能力两大核心难题,意义重大。介绍如下:


图源上海交大
钱璐璐(Lulu Qian),华裔科学家,现任加州理工学院生物工程教授。高考考入南京铁道医学院,后被东南大学合并,2002年毕业于东南大学,2007年获上海交通大学生物化学与分子生物学博士学位,师从贺林院士。2008年进入加州理工学院从事博士后研究,2011年1月至2013年6月任加州理工学院生物工程高级访问学者。主要研究方向为人工分子系统的设计与构建 。2012年2月-2012年11月,哈佛医学院访问学者。2013年-2018年,任加州理工学院助理教授;2019年-至今,教授。
2013年获美国国家科学基金会CAREER奖及日本大川奖。2019 年获得《美国化学会志》青年研究者奖,2019 年获得费曼纳米技术奖,2022 年获得施密特科学通才奖(Schmidt Science Polymath Award),2023 年获得理查德・费曼杰出教学奖(Feynman Prize for Excellence in Teaching)。
钱璐璐教授科研成果斐然,深受学生喜爱,获加州理工学院教学最高奖,此前在国内曾因3段婚姻引起争议。
重新定义分子学习的可能性
传统的分子计算系统面临着一个根本性的局限:它们只能执行预先编程的任务,无法像生物系统那样通过经验学习和适应环境。钱璐璐团队的DNA神经网络监督学习研究彻底打破了这一桎梏,首次实现了分子尺度的自主学习能力。
2025年9月3日,美国加州理工学院钱璐璐教授课题组在Nature上发表题为“Supervised learning in DNA neural networks”的研究性论文这项研究发现,DNA分子可以被编程为自主地在体外进行监督学习,系统学习从输入和期望响应的分子示例中执行模式分类。这为在从生物医学到软材料的各种物理系统中进行嵌入式学习和决策的分子机器打开了大门。
该论文作者仅2人, Kevin M. Cherry博士(2023-2024 博士后)为唯一第一作者,钱璐璐教授为唯一通讯作者。

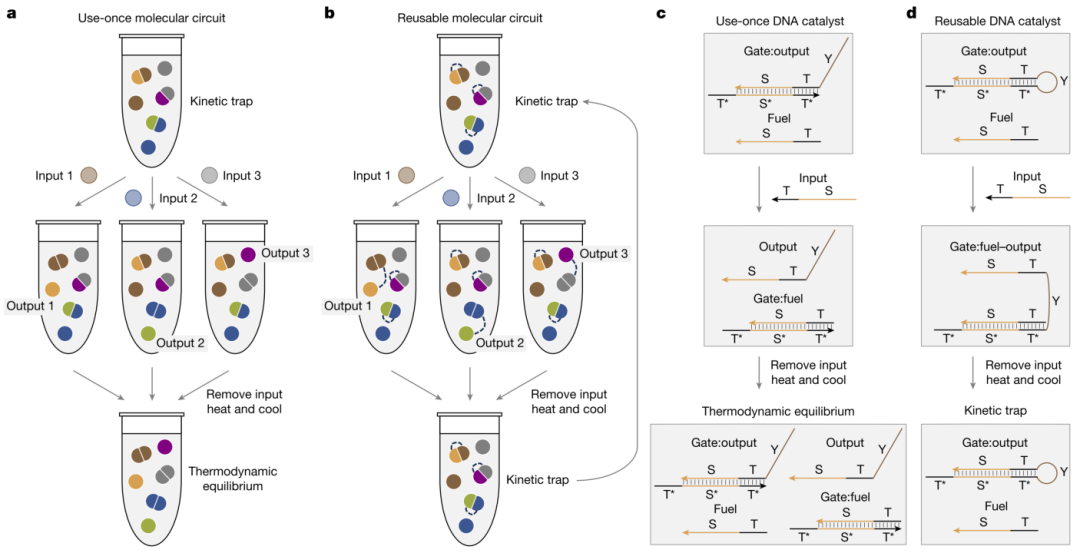
图1 | 热驱动可重用分子电路的概念。 a. 一次性分子电路。不同颜色的球代表不同单体,输入可催化解离二聚体并释放输出。 b. 可重用分子电路。虚线表示单体间的强柔性连接,热可复位系统至动力学陷阱状态。 c. 一次性DNA催化剂。 d. 可重用DNA催化剂。彩色线条代表DNA链,箭头表示3'端。
这一突破的核心在于两种全新的可激活门控结构的设计。可激活放大门控负责生成加权输入信号,能够精确地将输入位和记忆类别信息转化为可激活的权重;可激活转换门控则负责在训练过程中不可逆地生成激活剂,确保记忆的形成,同时又保留了与标签的可逆互作,避免单一训练模式的过度拟合。
这种设计使得DNA分子系统能够像生物神经网络一样,通过反复训练形成长期记忆,并将这种记忆应用于新的模式识别任务中。钱璐璐教授让大块大块的结构进行自我组装,形成完全自定义的结构。为了彰显这项技术的潜力,用 DNA 创造了世界上最小的“蒙娜丽莎”画。
钱璐璐教授团队创建世上最小版本《蒙娜丽莎的微笑》
实验结果令人震撼:该系统成功构建了包含700多个不同分子物种、超过1200个独特链段的100位记忆网络。在72个代表性测试中,系统展示了出色的分类功能,证明工程化的分子系统确实能够学习复杂的信息处理任务。更重要的是,这种学习能力具备了独立性、整合性、泛化性和稳定性四个关键特征,使其更接近于真正的人工细胞设想。
研究团队采用的"批训练"方法进一步验证了系统的鲁棒性。通过同时呈现同一类别的所有100位训练模式,系统能够形成清晰反映数字"0"和"1"形状的激活剂浓度分布。不同训练顺序下学习的结果保持一致,证明了该学习机制的可靠性和适应性。
热能驱动的可持续分子计算
在解决了学习能力问题之后,钱璐璐团队紧接着攻克了分子计算的另一个核心难题:能源供给的可持续性。
在人工分子机器领域,寻找一种像ATP或电力那样通用的能源一直是一个重大挑战。尽管DNA曾被用作燃料驱动纳米设备,但每个系统都需要不同的燃料序列,限制了其普适性。过去二十多年来,研究者们虽不断探索,却始终未能找到一种可持续的能源方式,使无酶分子电路能够反复执行复杂计算而不受废物积累的困扰。
据此,加州理工学院钱璐璐教授提出了一种突破性方案:利用热量为DNA逻辑电路和神经网络“充电”。研究表明,通过加热和冷却,可以使酶游离的DNA电路从热力学平衡状态恢复到非平衡的动力学陷阱状态,从而为后续计算提供能量。团队成功构建了包含超过200种分子物种的复杂逻辑电路与神经网络,系统可在几分钟内完成充电,并支持至少16轮连续计算,且无需担心废物积累导致的性能下降。这一策略为分子机器实现迭代计算、无监督学习等高级自主行为奠定了基础。
2025年10月1日,相关论文以“Heat-rechargeable computation in DNA logic circuits and neural networks”为题发表在Nature上。该论文作者仅2人, Tianqi Song博士(现任教职于UNC Greensboro助理教授)为唯一第一作者。钱璐璐教授为唯一通讯作者。

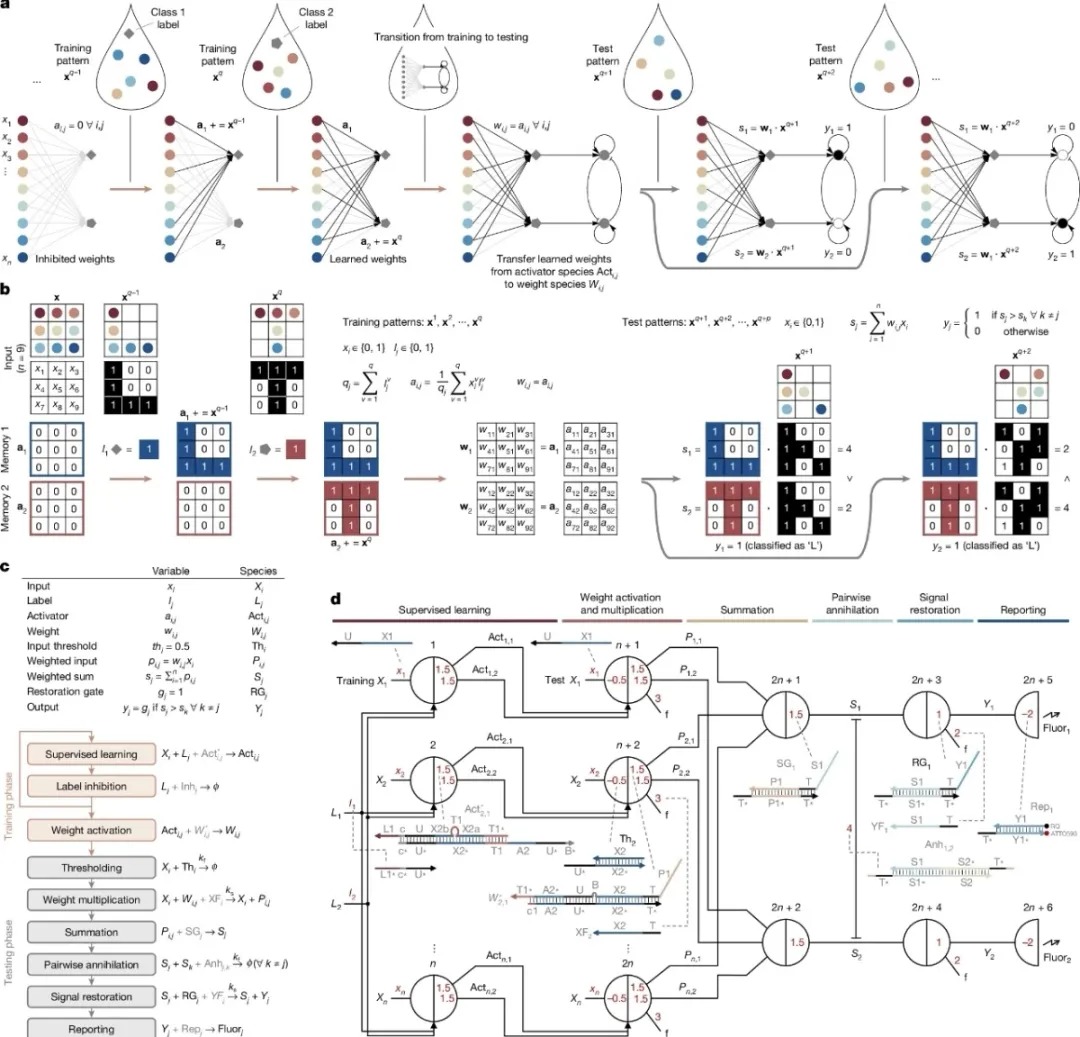
图1. DNA神经网络中监督学习的概念与设计
通过巧妙的分子设计,研究团队实现了酶游离DNA电路从热力学平衡状态恢复到非平衡动力学陷阱状态的过程,从而为后续计算提供所需能量。
这一技术的实际应用潜力巨大。团队成功构建了包含超过200种分子物种的复杂逻辑电路与神经网络,系统可在几分钟内完成充电,并支持至少16轮连续计算,且无需担心废物积累导致的性能下降。在一个七层DNA电路的验证实验中,该系统在连续16轮计算中表现出高度一致的性能,历经640小时与15次复位后仍能可靠响应。
更令人印象深刻的是100比特的双记忆"赢家通吃"神经网络的成功构建。该系统包含多达289种不同的DNA链,在单一试管中实现了213种分子的共存。即使在十轮连续测试后,网络仍能稳定分类不同输入模式,展现出优异的复位稳健性与系统兼容性。
迈向化学智能时代的战略意义
这两项研究的意义远超科学发现本身,它们代表了人类对智能本质理解的深化和对技术边界的拓展。从2006年Paul Rothemund开发DNA折纸术开始,DNA纳米技术已经走过了近二十年的发展历程。钱璐璐团队的工作标志着这一领域从静态结构设计向动态智能行为的历史性转变。
2006年3月16日,加州理工学院Paul Rothemund博士唯一作者在Nature上发表题为“ Folding DNA to create nanoscale shapes and patterns”研究并被选为当期封面,Paul Rothemund博士开发了一种“DNA折纸术”( DNA origami technique),像折叠一条长带子那样,把一条DNA长链反复折叠,形成需要的图形,就像用一根单线条绘制出整幅图画,这一技术也被戏称为“DNA 折纸”。
2017年12月7日,加州理工学院钱璐璐教授课题组开发了一种成本低廉的“DNA 折纸”技术,能让大块大块的结构进行自我组装,形成完全自定义的结构,用 DNA 创造了世界上最小的“蒙娜丽莎”画。相关研究以“Fractal assembly of micrometre-scale DNA origami arrays with arbitrary patterns”发表在Nature上,并登上当期 Nature 封面。

钱璐璐教授团队创建世上最小版本《蒙娜丽莎的微笑》

在生物医学应用方面,具备学习能力和可持续动力的DNA分子系统为精准医疗开辟了全新的可能性。这些系统可以在体内环境中自主学习病理模式,适应疾病的演变过程,并据此调整治疗策略。相比传统的药物递送系统,这种智能化的分子机器能够提供更加个性化和动态化的治疗方案。
在材料科学领域,热驱动的可充电分子计算为开发新一代智能材料奠定了基础。这些材料能够感知环境变化,学习优化响应策略,并在不同条件下表现出自适应的性能。从自愈合材料到环境响应型建筑材料,应用前景极为广阔。
更为重要的是,这些研究为人工智能的发展提供了全新的技术路径。与基于硅芯片的传统计算不同,分子计算具有超高的并行性、极低的能耗和独特的生物兼容性。随着摩尔定律逐渐接近物理极限,分子计算可能成为下一代信息技术的重要基础。钱璐璐团队的工作为实现真正的化学智能铺平了道路。
Lulu Qian Publications
Sarraf, Namita;Rodriguez, Kellen R. et al. (2023) Modular reconfiguration of DNA origami assemblies using tile displacementScience Robotics
Lapteva, Anna P.;Sarraf, Namita et al. (2022) DNA Strand-Displacement Temporal Logic CircuitsJournal of the American Chemical Society
Glynn, Allison T.;Davidson, Samuel R. et al. (2022) Developmental Self-Assembly of a DNA Ring with Stimulus-Responsive Size and Growth DirectionJournal of the American Chemical Society
Rodriguez, Kellen R.;Sarraf, Namita et al. (2021) A Loser-Take-All DNA CircuitACS Synthetic Biology
Taylor, Dallas N.;Davidson, Samuel R. et al. (2021) A Cooperative DNA CatalystJournal of the American Chemical Society
Clamons, Samuel;Qian, Lulu et al. (2020) Programming and simulating chemical reaction networks on a surfaceJournal of the Royal Society Interface
Tikhomirov, Grigory;Petersen, Philip et al. (2018) Triangular DNA Origami TilingsJournal of the American Chemical Society
Petersen, Philip;Tikhomirov, Grigory et al. (2018) Information-based autonomous reconfiguration in systems of interacting DNA nanostructuresNature Communications
Brijder, Robert;Qian, Lulu (2018) PrefaceNatural Computing
Cherry, Kevin M.;Qian, Lulu (2018) Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks Nature
For a complete publications list, see feeds.library.caltech.edu
Experimental work
[1] Neural network computation with DNA strand displacement cascades. Lulu Qian, Erik Winfree, and Jehoshua Bruck. Nature, 475:368-372, 2011.
[2] Scaling up digital circuit computation with DNA strand displacement cascades. Lulu Qian and Erik Winfree. Science, 332:1196-1201, 2011.
Theoretical work
[1] A simple DNA gate motif for synthesizing large-scale circuits. Lulu Qian and Erik Winfree. Journal of the Royal Society Interface, 8:1281-1297, 2011.
[2] Efficient Turing-universal computation with DNA polymers. Lulu Qian, David Soloveichik, and Erik Winfree. In Y. Sakakibara and Y. Mi, editors, DNA Computing and Molecular Programming, LNCS 6518:123-140, 2011.
论文链接:
https://www.nature.com/articles/nature24655
https://www.nature.com/articles/s41586-025-09570-2
https://www.nature.com/articles/s41586-025-09479-w
来源:科技头等舱、高分子科学、加州理工学院、科研大匠等,爱科会易仅用于学术交流,若相关内容侵权,请联系删除
近日,美国加州理工学院钱璐璐(Lulu Qian)教授课题组在2025年9月3日、10月1日分别以题为“Supervised learning in DNA neural networks”和“Heat-rechargeable computation in DNA logic circuits and neural networks” 发表2篇Nature研究论文。2篇论文作者都只有2人,钱璐璐教授是唯一通讯作者。2篇论文第一作者分别是Kevin M. Cherry博士(2023-2024 博士后),Tianqi Song 博士(现任教职于UNC Greensboro助理教授)。两项研究解决了分子计算领域长期存在的能源供给和学习能力两大核心难题,意义重大。介绍如下:


图源上海交大
钱璐璐(Lulu Qian),华裔科学家,现任加州理工学院生物工程教授。高考考入南京铁道医学院,后被东南大学合并,2002年毕业于东南大学,2007年获上海交通大学生物化学与分子生物学博士学位,师从贺林院士。2008年进入加州理工学院从事博士后研究,2011年1月至2013年6月任加州理工学院生物工程高级访问学者。主要研究方向为人工分子系统的设计与构建 。2012年2月-2012年11月,哈佛医学院访问学者。2013年-2018年,任加州理工学院助理教授;2019年-至今,教授。
2013年获美国国家科学基金会CAREER奖及日本大川奖。2019 年获得《美国化学会志》青年研究者奖,2019 年获得费曼纳米技术奖,2022 年获得施密特科学通才奖(Schmidt Science Polymath Award),2023 年获得理查德・费曼杰出教学奖(Feynman Prize for Excellence in Teaching)。
钱璐璐教授科研成果斐然,深受学生喜爱,获加州理工学院教学最高奖,此前在国内曾因3段婚姻引起争议。
重新定义分子学习的可能性
传统的分子计算系统面临着一个根本性的局限:它们只能执行预先编程的任务,无法像生物系统那样通过经验学习和适应环境。钱璐璐团队的DNA神经网络监督学习研究彻底打破了这一桎梏,首次实现了分子尺度的自主学习能力。
2025年9月3日,美国加州理工学院钱璐璐教授课题组在Nature上发表题为“Supervised learning in DNA neural networks”的研究性论文这项研究发现,DNA分子可以被编程为自主地在体外进行监督学习,系统学习从输入和期望响应的分子示例中执行模式分类。这为在从生物医学到软材料的各种物理系统中进行嵌入式学习和决策的分子机器打开了大门。
该论文作者仅2人, Kevin M. Cherry博士(2023-2024 博士后)为唯一第一作者,钱璐璐教授为唯一通讯作者。


图1 | 热驱动可重用分子电路的概念。 a. 一次性分子电路。不同颜色的球代表不同单体,输入可催化解离二聚体并释放输出。 b. 可重用分子电路。虚线表示单体间的强柔性连接,热可复位系统至动力学陷阱状态。 c. 一次性DNA催化剂。 d. 可重用DNA催化剂。彩色线条代表DNA链,箭头表示3'端。
这一突破的核心在于两种全新的可激活门控结构的设计。可激活放大门控负责生成加权输入信号,能够精确地将输入位和记忆类别信息转化为可激活的权重;可激活转换门控则负责在训练过程中不可逆地生成激活剂,确保记忆的形成,同时又保留了与标签的可逆互作,避免单一训练模式的过度拟合。
这种设计使得DNA分子系统能够像生物神经网络一样,通过反复训练形成长期记忆,并将这种记忆应用于新的模式识别任务中。钱璐璐教授让大块大块的结构进行自我组装,形成完全自定义的结构。为了彰显这项技术的潜力,用 DNA 创造了世界上最小的“蒙娜丽莎”画。
钱璐璐教授团队创建世上最小版本《蒙娜丽莎的微笑》
实验结果令人震撼:该系统成功构建了包含700多个不同分子物种、超过1200个独特链段的100位记忆网络。在72个代表性测试中,系统展示了出色的分类功能,证明工程化的分子系统确实能够学习复杂的信息处理任务。更重要的是,这种学习能力具备了独立性、整合性、泛化性和稳定性四个关键特征,使其更接近于真正的人工细胞设想。
研究团队采用的"批训练"方法进一步验证了系统的鲁棒性。通过同时呈现同一类别的所有100位训练模式,系统能够形成清晰反映数字"0"和"1"形状的激活剂浓度分布。不同训练顺序下学习的结果保持一致,证明了该学习机制的可靠性和适应性。
热能驱动的可持续分子计算
在解决了学习能力问题之后,钱璐璐团队紧接着攻克了分子计算的另一个核心难题:能源供给的可持续性。
在人工分子机器领域,寻找一种像ATP或电力那样通用的能源一直是一个重大挑战。尽管DNA曾被用作燃料驱动纳米设备,但每个系统都需要不同的燃料序列,限制了其普适性。过去二十多年来,研究者们虽不断探索,却始终未能找到一种可持续的能源方式,使无酶分子电路能够反复执行复杂计算而不受废物积累的困扰。
据此,加州理工学院钱璐璐教授提出了一种突破性方案:利用热量为DNA逻辑电路和神经网络“充电”。研究表明,通过加热和冷却,可以使酶游离的DNA电路从热力学平衡状态恢复到非平衡的动力学陷阱状态,从而为后续计算提供能量。团队成功构建了包含超过200种分子物种的复杂逻辑电路与神经网络,系统可在几分钟内完成充电,并支持至少16轮连续计算,且无需担心废物积累导致的性能下降。这一策略为分子机器实现迭代计算、无监督学习等高级自主行为奠定了基础。
2025年10月1日,相关论文以“Heat-rechargeable computation in DNA logic circuits and neural networks”为题发表在Nature上。该论文作者仅2人, Tianqi Song博士(现任教职于UNC Greensboro助理教授)为唯一第一作者。钱璐璐教授为唯一通讯作者。


图1. DNA神经网络中监督学习的概念与设计
通过巧妙的分子设计,研究团队实现了酶游离DNA电路从热力学平衡状态恢复到非平衡动力学陷阱状态的过程,从而为后续计算提供所需能量。
这一技术的实际应用潜力巨大。团队成功构建了包含超过200种分子物种的复杂逻辑电路与神经网络,系统可在几分钟内完成充电,并支持至少16轮连续计算,且无需担心废物积累导致的性能下降。在一个七层DNA电路的验证实验中,该系统在连续16轮计算中表现出高度一致的性能,历经640小时与15次复位后仍能可靠响应。
更令人印象深刻的是100比特的双记忆"赢家通吃"神经网络的成功构建。该系统包含多达289种不同的DNA链,在单一试管中实现了213种分子的共存。即使在十轮连续测试后,网络仍能稳定分类不同输入模式,展现出优异的复位稳健性与系统兼容性。
迈向化学智能时代的战略意义
这两项研究的意义远超科学发现本身,它们代表了人类对智能本质理解的深化和对技术边界的拓展。从2006年Paul Rothemund开发DNA折纸术开始,DNA纳米技术已经走过了近二十年的发展历程。钱璐璐团队的工作标志着这一领域从静态结构设计向动态智能行为的历史性转变。
2006年3月16日,加州理工学院Paul Rothemund博士唯一作者在Nature上发表题为“ Folding DNA to create nanoscale shapes and patterns”研究并被选为当期封面,Paul Rothemund博士开发了一种“DNA折纸术”( DNA origami technique),像折叠一条长带子那样,把一条DNA长链反复折叠,形成需要的图形,就像用一根单线条绘制出整幅图画,这一技术也被戏称为“DNA 折纸”。
2017年12月7日,加州理工学院钱璐璐教授课题组开发了一种成本低廉的“DNA 折纸”技术,能让大块大块的结构进行自我组装,形成完全自定义的结构,用 DNA 创造了世界上最小的“蒙娜丽莎”画。相关研究以“Fractal assembly of micrometre-scale DNA origami arrays with arbitrary patterns”发表在Nature上,并登上当期 Nature 封面。

钱璐璐教授团队创建世上最小版本《蒙娜丽莎的微笑》

在生物医学应用方面,具备学习能力和可持续动力的DNA分子系统为精准医疗开辟了全新的可能性。这些系统可以在体内环境中自主学习病理模式,适应疾病的演变过程,并据此调整治疗策略。相比传统的药物递送系统,这种智能化的分子机器能够提供更加个性化和动态化的治疗方案。
在材料科学领域,热驱动的可充电分子计算为开发新一代智能材料奠定了基础。这些材料能够感知环境变化,学习优化响应策略,并在不同条件下表现出自适应的性能。从自愈合材料到环境响应型建筑材料,应用前景极为广阔。
更为重要的是,这些研究为人工智能的发展提供了全新的技术路径。与基于硅芯片的传统计算不同,分子计算具有超高的并行性、极低的能耗和独特的生物兼容性。随着摩尔定律逐渐接近物理极限,分子计算可能成为下一代信息技术的重要基础。钱璐璐团队的工作为实现真正的化学智能铺平了道路。
Lulu Qian Publications
Sarraf, Namita;Rodriguez, Kellen R. et al. (2023) Modular reconfiguration of DNA origami assemblies using tile displacementScience Robotics
Lapteva, Anna P.;Sarraf, Namita et al. (2022) DNA Strand-Displacement Temporal Logic CircuitsJournal of the American Chemical Society
Glynn, Allison T.;Davidson, Samuel R. et al. (2022) Developmental Self-Assembly of a DNA Ring with Stimulus-Responsive Size and Growth DirectionJournal of the American Chemical Society
Rodriguez, Kellen R.;Sarraf, Namita et al. (2021) A Loser-Take-All DNA CircuitACS Synthetic Biology
Taylor, Dallas N.;Davidson, Samuel R. et al. (2021) A Cooperative DNA CatalystJournal of the American Chemical Society
Clamons, Samuel;Qian, Lulu et al. (2020) Programming and simulating chemical reaction networks on a surfaceJournal of the Royal Society Interface
Tikhomirov, Grigory;Petersen, Philip et al. (2018) Triangular DNA Origami TilingsJournal of the American Chemical Society
Petersen, Philip;Tikhomirov, Grigory et al. (2018) Information-based autonomous reconfiguration in systems of interacting DNA nanostructuresNature Communications
Brijder, Robert;Qian, Lulu (2018) PrefaceNatural Computing
Cherry, Kevin M.;Qian, Lulu (2018) Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks Nature
For a complete publications list, see feeds.library.caltech.edu
Experimental work
[1] Neural network computation with DNA strand displacement cascades. Lulu Qian, Erik Winfree, and Jehoshua Bruck. Nature, 475:368-372, 2011.
[2] Scaling up digital circuit computation with DNA strand displacement cascades. Lulu Qian and Erik Winfree. Science, 332:1196-1201, 2011.
Theoretical work
[1] A simple DNA gate motif for synthesizing large-scale circuits. Lulu Qian and Erik Winfree. Journal of the Royal Society Interface, 8:1281-1297, 2011.
[2] Efficient Turing-universal computation with DNA polymers. Lulu Qian, David Soloveichik, and Erik Winfree. In Y. Sakakibara and Y. Mi, editors, DNA Computing and Molecular Programming, LNCS 6518:123-140, 2011.
论文链接:
https://www.nature.com/articles/nature24655
https://www.nature.com/articles/s41586-025-09570-2
https://www.nature.com/articles/s41586-025-09479-w
来源:科技头等舱、高分子科学、加州理工学院、科研大匠等,爱科会易仅用于学术交流,若相关内容侵权,请联系删除

2026.02.28 - 2026.03.02 中国 成都

2026.02.06 - 2026.02.08 日本 大阪

2026.02.04 - 2026.02.06 泰国 普吉岛

2026.02.28 - 2026.03.02
中国 成都
投稿截止 2025.11.30

2026.02.06 - 2026.02.08
日本 大阪
投稿截止 2025.12.05

2026.02.04 - 2026.02.06
泰国 普吉岛
投稿截止 2025.11.25